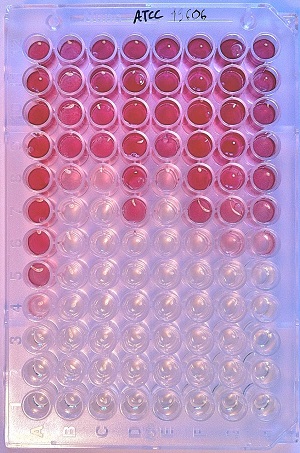
BAC-SCREEN
Bacteria within the reach of molecules
BAC-SCREEN est une plateforme spécialisée dans le criblage de molécules sur bactéries (high content).
Créée en juin 2011, sous la co-tutelle d’Aix Marseille Université et de l’Institut de Recherche Biomédicale des Armées, elle est rattachée au laboratoire MCT (Membranes et cibles Thérapeutiques) situé sur le campus Timone, Marseille.


BAC-SCREEN est labellisée « Plateforme Technologique du site d'Aix-Marseille ».
Elle appartient au réseau des plateformes technologiques impliquées dans les approches de chemobiologie, ChemBioScreen (GDR CNRS 3735).
La plateforme a pour vocation première l'identification de nouveaux agents antibactériens ou d'adjuvants potentialisateurs de l'activité d'antibiotiques existants et la compréhension de leurs mécanismes d'action.
Elle est ouverte aux industriels et aux académiques, et fonctionne par prestations de service ou projets collaboratifs.
BAC-SCREEN propose son expérience et son savoir-faire à plusieurs niveaux :
- Le criblage in cellulo de chimiothèques :
° Détermination de la sensibilité bactérienne par microdilution en milieu liquide.
° Etude des interactions entre composés et antibiotiques/biocides.
- L'optimisation de molécules Hits dans une stratégie de "Hit to lead" en collaboration avec les chimistes de l'Unité.
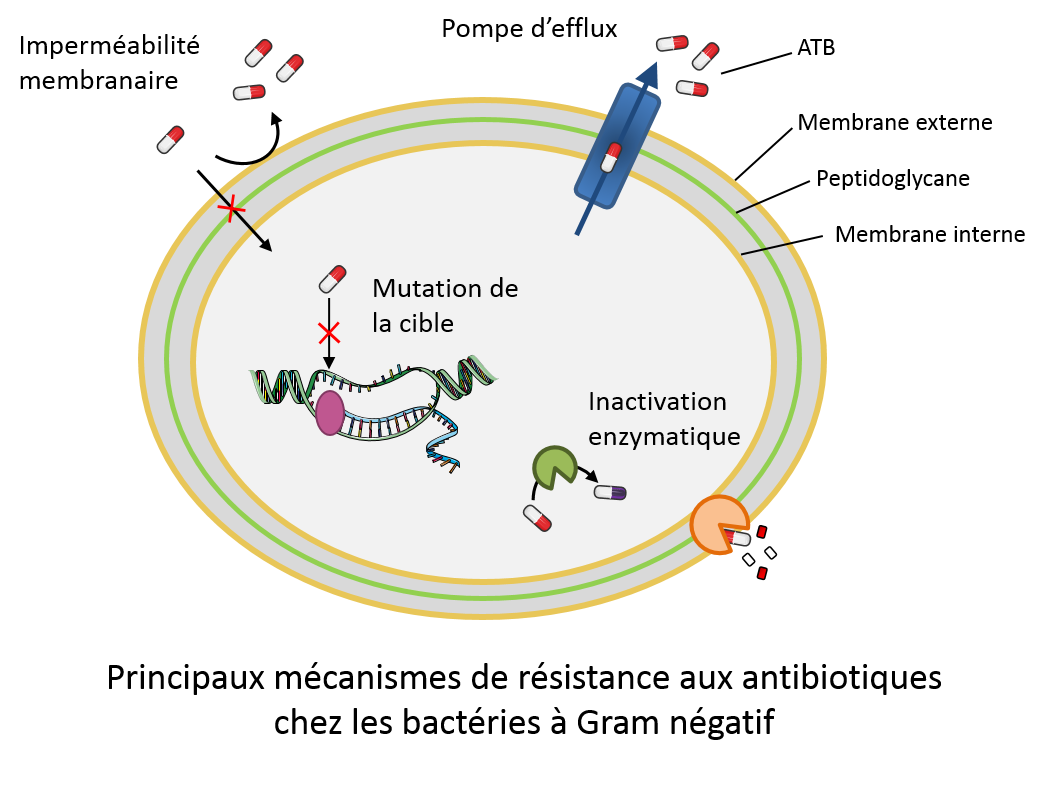
- Le développement de tests d'activité en relation avec l'intégrité de la membrane bactérienne et les phénomènes d'efflux actifs :
° Mesure de la capacité de composés à perméalibiliser les membranes bactériennes et à diffuser à travers les membranes bactériennes via les porines.
° Mesure de l'activité de composés sur le potentiel de membrane.
° Mesure de l'activité d'inhibiteur de pompes d'efflux.
° Mesure de la capacité de composés à inhiber la formation et le développement de biofilms.
- L'accès à la souchothèque :
° Souches de référence et isolats cliniques des principales bactéries pathogènes chez l'homme et chez les animaux.
° Mutants, surproducteurs de mécanismes de résistance ou à l'inverse mutés sélectivement par délétion de certains de ces mécanismes.
- L'accès à des chimiothèques :
° Issues de collaborations académiques ou préparées par les chimistes de l'Unité.
° Issues de collaborations industrielles.
- Le développement de nouvelles technoloqies (essais, prototypage, preuve de concept).
Environnement de Sécurité Microbiologique de Niveau 2
BAC-SCREEN peut prendre en charge des études sur toutes les bactéries exigeant un confinement microbiologique de niveau 2.
C’est une structure évolutive qui a la volonté de développer et d’acquérir de nouvelles méthodes pour répondre à la demande.
BAC-SCREEN accorde une grande importance à la formation
L’accueil de doctorants fait partie intégrante de l’activité de la plateforme et de sa stratégie de développement, basée sur le mode collaboratif d’apport mutuel de compétences.
Vincent Lôme, doctorant qui a travaillé pour la structure, a par exemple développé une méthode originale permettant le criblage de nouveaux agents.
« Cette méthode permet la détection spécifique de chimiosensibilisants tout en décrivant leur mécanisme d'action : inhibiteur d'efflux ou agent perméabilisant. Cette approche repose sur la comparaison des éléments d'une chimiothèque dans un cadre homogène, par la réalisation robotisée des tests associée à un traitement des données automatisé et appliqué systématiquement. Une banque de quatre-vingts composés, préalablement caractérisés dans la littérature, a été assemblée et testée afin de valider la méthode proposée. Les outils développés permettront d'explorer de nouveaux espaces chimiques et serviront de support pour les campagnes d'amélioration par pharmacomodulations (i.e. drug-design). » Vincent Lôme, doctorant.
Équipements spécifiques
-
1 FREEDOM EVO 150 : automate de pipetage équipé en aiguilles ou cônes jetables.
-
1 Infinite M200 Pro : lecteurs de microplaques (absorbance UV-visible, Fluorescence, luminescence), collecte des résultats.
-
1 LIMS (Laboratory Information Management System) : système de gestion informatisé de la souchothèque, des chimiothèques et des plaques d'expérience. Gestion des stocks, analyse des résultats, comptes rendus.
-
2 Congélateurs -80°C : stockage des souches bactériennes.
-
1 Sunrise : lecteur de microplaques (absorbance UV-visible thermostaté).
-
9 Incubateurs : enceintes de culture de bactéries à température contrôlée.



Collaborations
Depuis 2011, BAC-SCREEN participe à plusieurs projets :
Programme IMI-Translocation (contrat européen) qui a vocation à étudier le transport membranaire des antibiotiques.
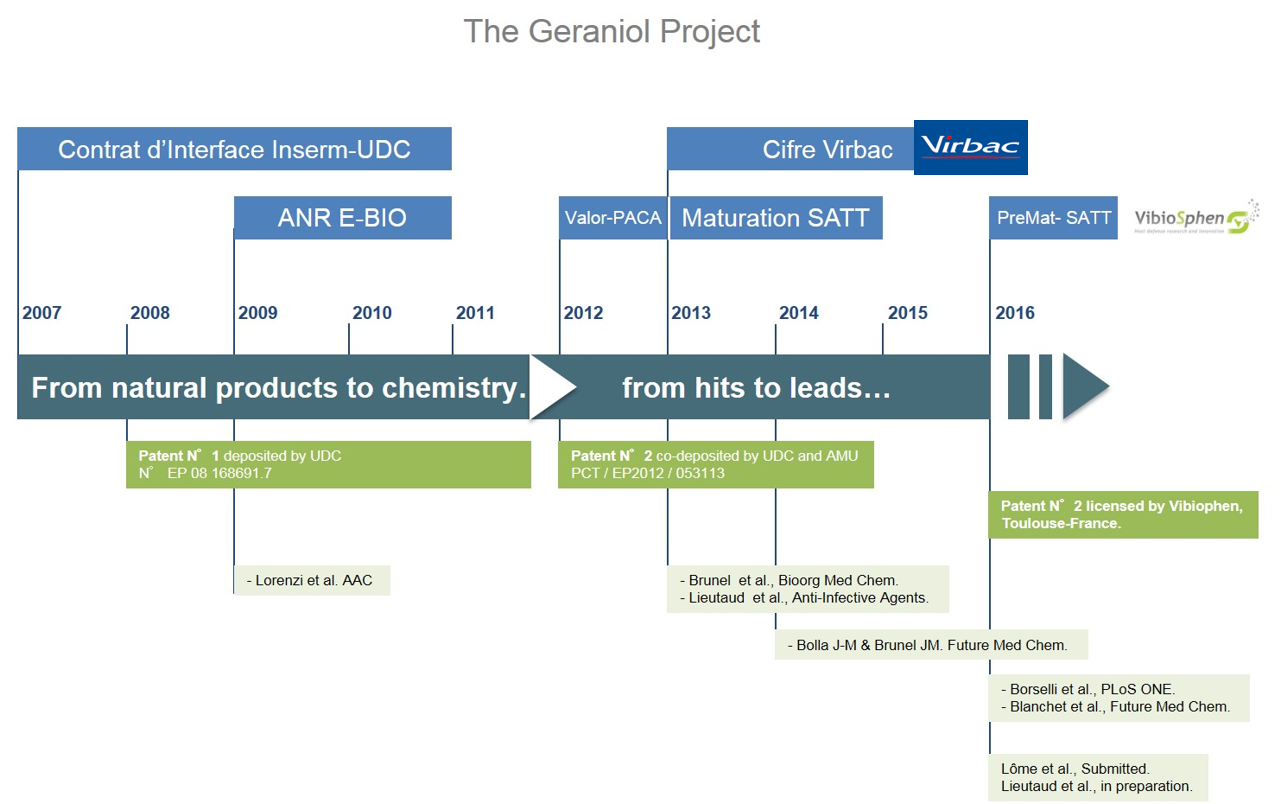
Projet Géraniol (SATT Sud-Est, CIFRE) : recherche d’un nouvel adjuvant.
Projet "RECHAP" (A*MIDEX Pépinière d'Excellence - 2019-2020) qui vise à démontrer l'efficacité d'action de formulations associant souches bactériennes probiotiques et surfactants naturels d'origine microbienne sur la colonisation de surface par des bactéries pathogènes et la dégradation de biofilms.
Par ailleurs, la plateforme collabore avec des laboratoires académiques du CiNaM, le Laboratoire de Pharmacognosie-Ethnopharmacologie-Homéopathie d'Aix-Marseille Université (Marseille, France), l'université Catholique de Louvain (Belgique), l'Institut de chimie Organique de Budapest (Hongrie), l'ISPB (Lyon), l'université Jagellone (Cracovie, Pologne), l'université de Lyon I (Lyon, France : Dr L. Rocheblave / Pr. K. Kononowicz).
Autres collaborations (biotechnologie) :